Minbiziarekin lotutako mutazioen efektua aurreikusteko software bioinformatikoa sortu du UPV/EHUko diziplinarteko talde batek. Softwarea minutu bakarrean 40.000 proteina aztertzeko gai da eta, besteak beste, minbizia bezalako gaixotasunak eragin ditzaketen proteina mutazioak aztertzeko aukera ematen du.
Biologiak eta informatikak bat egin dute minbizian eragin ditzaketen proteina mutazioak aztertzen dituen software bat sortzeko. Asier Fullaondo eta José Antonio Rodríguez genetistek eta Gorka Prieto telekomunikazioetako ingeniariak WREGEX 2.0 sortu dute, 40.000 proteinaren informazioa minutu batean aztertu eta konbina dezakeen aplikazio bioinformatiko askea, erraza, moldakorra eta, batik bat, bizkorra.
Proteinak aminoazidoen kateek osatzen dituzte, eta proteina bakoitzak funtzio diskretua duten aminoazido sekuentzia laburrak ditu, motibo funtzional esaten zaienak. Motibo horietako batzuk zehaztuta egoten dira jada, eta beste batzuk, berriz, zehaztu gabe. Motibo funtzional bat aldatuta agertzen bada, litekeena da mutazio horrek minbizia bezalako gaixotasunen bat eragitea. Proteina baten funtzioa ikertzeko, hasteko, proteina horrek izan ditzakeen aldaketak egiaztatu behar dira. Kontuan hartuta giza proteomaren egungo zirriborroa 40.000 proteina baino gehiagoz osatuta dago, proteina bakoitzaren mutazioak bilatzea egundoko lana da.
Hori dela eta, hiru ikertzaileak tresna bioinformatikoa lantzen hasi ziren: José Antonio Rodríguezek galdera biologikoa planteatu zuen; Asier Fullaondo, berriz, tresna eta datu-base bioinformatikoen gaineko ezagutzaz baliatu zen; eta Gorka Prietok, azkenik, programatzeko gaitasuna aplikatu zuen.
Hasieran, doktoreek motibo funtzionalak (proteina baten zeregin espezifikoak gauzatzen dituzten aminoazido talde txikiak) automatikoki aurreikusteko eta bilatzeko software bat garatu zuten (WREGEX). Hala, programa probatu zuten, proteina bat zelularen nukleotik zitoplasmaraino eramaten duten motiboak aurreikusteko, hots, esportazio nuklearreko seinaleak deiturikoak. Ikerketa fase hori bukatzean, 2014an, artikulu bat kaleratu zuten Bioinformatics aldizkarian. Baina, José Antonio Rodríguezek dioen moduan, “ikerkuntzan, galdera baten erantzunak are galdera gehiago ekartzen ditu”. Eta galdera hauxe sortu zitzaien: zer proteinak izan dezakete beren aminoazido sekuentzian minbizira mutaturiko motibo funtzional bat?
Taldeak beste urrats bat egin, eta COSMIC (Catalogue of Somatic Mutations in Cancer) katalogoan jasota dauden giza proteina guztien sekuentziari buruzko informazioa konbinatu zuen (katalogo horrek, hain zuen ere, minbiziarekin zerikusia duten mutazioak biltzen ditu). Modu horretan, beste bertsio bat sortu zuten (WREGEX 2.0), proteina normala eta mutatua alderatzeko eta, horrela, aldatu diren eta minbiziarekin lotura izan dezaketen motibo funtzionalak aurreikusi ahal izateko. “Era berean, baliteke zuk esperientzia izatea motibo baten funtzionamenduan eta jakin nahi izatea zer proteinatan ager daitekeen edo ea minbizira alda daitekeen. Bada, software honekin, ikertzen hasteko proteinak lor ditzakezu”, azaldu du Gorka Prietok.
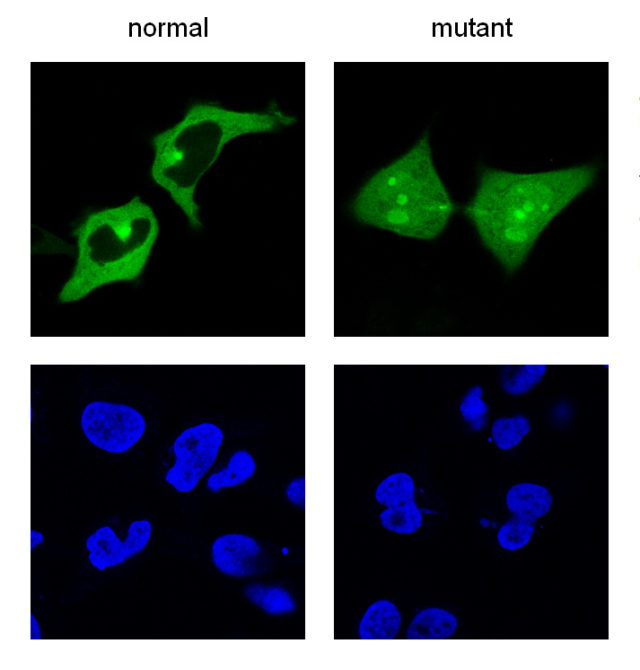
Behin programa bioinformatikoa garatutakoan, probatzeko garaia iritsi zitzaien; horretarako, zeluletan esportatzeko saiakuntza bat egin zuten. Hala, berriz ere, proteina zelularen nukleotik kanpora mugitzeko motiboa izan zezaketen zenbait proteina hautatu zituzten. Haien funtzionamendua aztertu, eta, COSMIC katalogoan deskribatutako tumore mutazioen arabera aldatu ondoren, berriro egin zuten saiakuntza. Horrela, egiaztatu ahal izan zuten hautaturiko proteinek esportazio seinale gisa jokatzen zutela, mutazioak funtzionamenduari eragiten ziola, eta, horrenbestez, softwarea baliagarria zela.
Tresnak hiru informazio mota konbinatzen ditu: proteinen sekuentziak, motibo funtzionalak eta minbiziaren mutazioak. “WREGEX 2.0 aplikazioaren ezaugarri nagusietako bat da aldi berean azter ditzakeela proteoma oso konplexuak, proteina mordoa dutenak, eta minbiziaren mutazioekin konbina dezakeela informazioa, saiakuntzaren kasuan bezala. Dena dela, beste mutazio mota batzuei buruzko informazioa duten datu baseak ere erabil ditzakegu. Abantaila bat du, gainera: minutu batean 40.000 proteina aztertzeko gai da, eta beste analisi programa batzuekin, berriz, zenbait minutu behar dira proteina bakarra aztertzeko”, azaldu du Asier Fullaondok. Horrenbestez, software horren bidez aurreikus daiteke proteina baten asalduak gaixotasuna eragin dezakeela, eta ez soilik minbizia.
Erreferentzia bibliografikoa:
Gorka Prieto, Asier Fullaondo & Jose A. Rodríguez. Proteome-wide search for functional motifs altered in tumors: Prediction of nuclear export signals inactivated by cancer-related mutations. Scientific Reports. doi:10.1038/srep25869
Iturria:
UPV/EHUko komunikazio bulegoa: Minbiziarekin lotutako mutazioen efektua aurreikusteko software bioinformatikoa.