Masa-espektrometria (MS) orain 100 urtetik kimikarientzako ohiko teknika izan arren, azken 3 hamarkadetan baino ez da erabili proteinak analizatzeko. Edozein analito MSz analizatu nahi badugu, berau ionizatu eta gas fasera aldatu behar da.
Proteinak eta proteina zatikiak (peptidoak) bezalako makromolekulak ionizatzea eta gas fasera aldatzea elefanteak hegaz egitea hainako egitekotzat jo zen luzaroan. Ionizazio “arina” izena eman zitzaien bi ionizazio-hurbilketa garatu eta komertzializatu zirenean, baina egingarri bilakatu zen proteinen tamainako makromolekula handiak analizatzea, bai soluzio likido batean, baita egoera lehor-kristalino batean ere; bi ionizazio mota horiei electrospray ionizazioa (ESI) eta matrix assisted laser desorption ionizazioa (MALDI) deritze, hurrenez hurren. ESIn, analitoa duen soluzioari tentsio altua eragiten zaio zulodun orratz estu batetik igarotzerakoan. Kargatutako molekula-soluzioa lurrundu ahala, sortutako tantatxoen tamaina txikituz eta txikituz doa gas fasera iritsi arte pasatzen den arte. MALDIn berriz, laserra disparatzen zaio altzairuzko plater bati. Bertan, analitoa matrize batekin batera dago lehortuta. Matrizeak laserraren energia xurgatzen du eta energia hori laginari transferitzen diolarik, gas fasera aldarazten du.
Baina, zergatik proteinak? Zergatik hainbesteko jakin-mina proteinak direla eta? interesa proteinak aztertzeko? Bada bizidun ororen oinarrizko molekulak direlako. Proteinak dira gure zeluletako ataza desberdinak betetzen dituzten langile finak; besteak beste, erreakzio kimikoak katalizatu, molekulak garraiatu, eta zelulen egitura mantentzen dute, edota patogenoen aurka babesten gaituzte proteinek. Gure gorputza trilioika zelulez osatuta dago, eta zelula bakoitzak milaka proteina desberdin izan ditzake. Hain zuzen ere, zelula mota bakoitzak proteina katalogo edo sorta jakina duelako, funtzio eta ezaugarri bereziak ditu. Bizidun baten zelula guztietan dagoen gene-informazioa, hots, genoma berdin-berdina da. Geneetan dago idatzita proteina bakoitza ekoizteko informazioa. Bestela esanda, geneak proteinak egiteko errezetak dira, eta errezeta horietako bakoitzaren arabera lortzen da proteina bat edo beste bat.
Zelula batek une jakin batean duen proteina-bildumari proteoma deritzo. Genoma ez bezala, zeluletako proteoma etengabe aldatzen da; zeluletara heltzen den edozein estimulu edo erasori aurre egiteko, alegia, ingurunera egokitzeko, proteina-konbinazio desberdinak sortu, eta arituko dira elkarlanean. Horrela uler daiteke, genoma bakarra izanik, beldar bat nola bilakatu daitekeen tximeleta (1. irudia.)

Zeluletako proteinek, gure antzera, euren “sare sozialak” dituztela esan dezakegu. Bakarrik baino elkarlanean betetzen dituzte beraien funtzioak. Halere, elkarrekintza horiek, gehienetan, iragankorrak izaten dira, zelularen beharren arabera sortu eta desegiten direlako. Milioika proteina-molekula izanik, gure planetan bizi garen gizaki guztien artekoa bezain konplexua da zelula bakar bateko elkarrekintza-sarea. Proteina-sare horien funtzionamenduak baldintzatuko du zelularen osasun maila eta, ondorioz, organismo osoaren osasun maila ere. Horregatik da hain garrantzitsua proteinak eta euren elkarrekintzak, alegia proteomak ikertzea eta osotasunean ulertzen saiatzea. Hori horrela izanik, gaur egungo biologiaren funtsezko erronketako bat bilakatu da proteomak kualitatiboki eta kuantitatiboki deskribatzea. Erronka horri aurre egiteko, proteomikak hartu duen gidaritza inork gutxik jartzen du zalantzatan.
Proteomikak proteomak ikertzen ditu. 1975. urtean lehenengoz Escherichia coli bakterioaren milaka proteina banatu ziren bi dimentsioko gel-elektroforesiaren bidez (2DE). Banatutako proteinak identifikatzea, hots, zeintzuk ziren zehaztea, bestelako erronka bat zen; erronka horri heltzeko ezinbestekoa izan zen alde batetik, sekuentziazio partziala egiteko tekniken garapena, Edman degradazioa esaterako. Bestetik, MS teknika bera proteinen analisirako doitzea.
Egun, MS da proteinak identifikatzeko ohiko metodoa. Proteomak osotasunean ulertzeko baina, identifikatzeaz gain, kuantifikatu egin behar dira proteinak. Hori egiteko lehen urratsak 2DE gelen irudi-analisien bidez eman ziren. Gaur egun azterketa proteomikoetarako teknika egokia izaten jarraitzen badu ere, leku txikia hartzen du masa-espektrometrian oinarritutako proteomikaren aldean (MS-based proteomics). Izan ere, proteina-lagin konplexuak analizatzeko, hots, proteinak identifikatzeko eta kuantifikatzeko, MS da aukerako metodoa gaur-gaurkoz. Izan ere, ezinbesteko teknika bilakatu da, genometan kodetuta dagoen informazioa interpretatzeko orduan.
Arestian esan bezala, ESI eta MALDI dira proteinak edota peptidoak ionizatu eta hegaz eginarazteko ohiko teknikak. MALDI-MS teknika peptido-nahastura nahiko bakunak analizatzeko erabili ohi da, baina ESI-MS sistemak (LC-MS, liquid chromatography-MS) dira lagin konplexuak analizatzeko egokienak.
Proteomak berez konplexuak izanik, LC-MS teknikak dira proteomikan gailendu direnak. Izan ere, LC-MSaren masa-zehaztasuna, sentikortasuna, bereizmena, eta abiadura dira teknikaren arrakastaren gako. Azken urteotan, LC-MS prozedura ezberdinak erabilita makina bat proteoma identifikatu, kuantifikatu eta karakterizatu dira. Proteomikaren lorpenen artean nabarmentzekoak dira 2014an argitaratutako giza proteomaren bi zirriborro, eta gaur egun Giza Proteomaren Erakundea (Human Proteome Organization, HUPO) gidatzen ari den Giza Proteomaren Proiektua (Human Proteome Project, HPP).
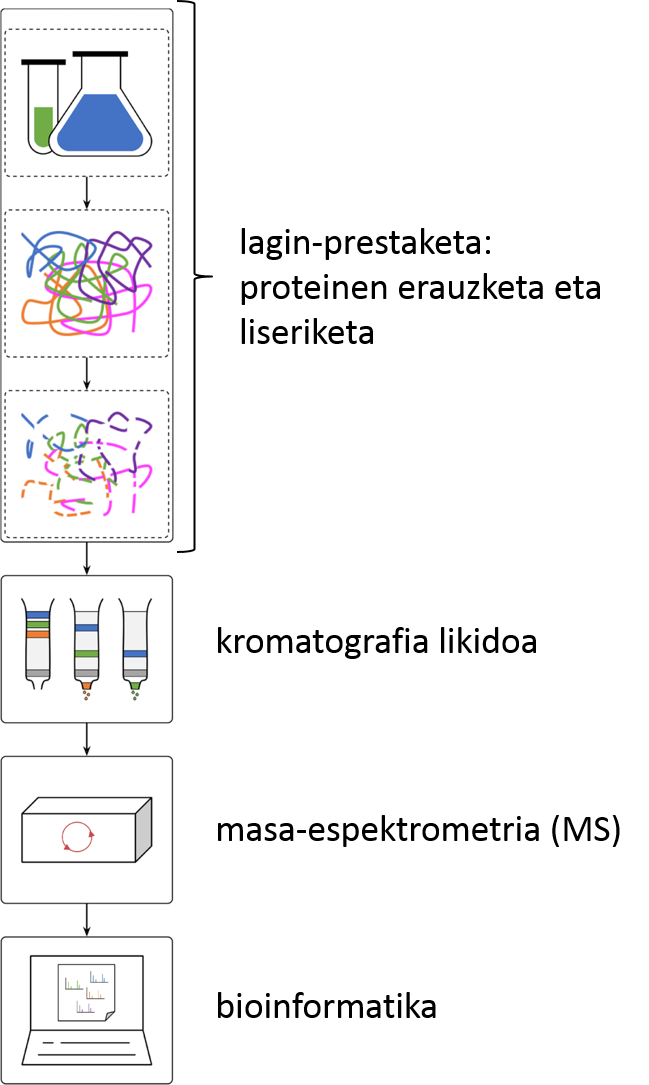
Baina zer da zehazki proteomikako laborategi batean egiten duguna? Edozein delarik ere eskuetan dugun lagina (biopsiaren bateko lagin bat, animalia edo landare-zelulak, bakterioak, jariakin biologikoren bat…), berau analizatzeko erabili ohi dugun lan-fluxua bottom-up proteomika izenekoa da (2. irudia). Lehenik eta behin, laginetik proteinak erauzten ditugu. Ondoren, proteinak liseritzen dituen entzima bat erabiliz, tripsina izaten da gehien bat, proteinak peptido izeneko zatikitan txikitzen ditugu. Peptido-nahasketa elektrospray ionizazioari akoplatutako alderantzizko faseko kromatografia likidoan banatuko dira. Zutabetik eluitu ahala, peptidoak ionizatu, eta masa-espektrometroan sartuko dira. Bertan, peptido-ioiak masa-karga (m/z) erlazioaren arabera banatu eta detektatuko dira. Peptido ugarienak gainontzekoetatik isolatuko dira, eta zatikatu egingo dira fragmentazio-espektroak osatzeko. Espektro horiek guztiak dira peptidoak identifikatu eta kuantifikatzeko informazioa gordetzen dutenak. Azkenik, masa-espektrometroak jasotako datu guztiak konputazio-erraminta berezien bidez analizatuko dira. Finean, jakingo dugu guk aztertutako laginean dauden proteinak zeintzuk diren, eta zein kopurutan agertzen diren.
Azken hamarkadan, MSean oinarritutako proteomika biozientzietarako analisi-tresna nagusienetakoa bilakatu da. Izan ere, proteinak ertz askotatik ikertzea ahalbidetu digu, eta euren egiturak, aldaerak, kopuruak, itzulpen osteko eraldaketak, eta elkarrekintzak ezagutzeko bide eman digu. Horrez gain, proteomikari esker, zehaztu ahal izango dugu zeintzuk diren zenbait gaixotasunen garapenean zerikusia duten proteinak, hala nola, gaixotasunen diagnostikoa egiteko baliagarriak izango diren biomarkatzaileak. Datozen urteetan, zelulen baldintza ezberdinetako proteina katalogoak osatuz joango gara. Genomika, epigenomika, metabolomika eta eskala handian egiten diren bestelako “omikatan” lortutako emaitzekin batera, proteomikaren eskutik heldutako aurkikuntzek zelula ereduak gauzatzen lagunduko digute. Izan ere, hori da joera: diziplinarteko ezagutzak uztartuz, prozesu biologikoen konplexutasuna argitzen lagunduko diguten matematika eta estatistika-ereduak lortzea; horretan ere, proteomikak zeresana emango du.
Erreferentzia bibliografikoak
- Fenn JB (2003) “Electrospray wings for molecular elephants (Nobel lecture)” Angew Chem Int Ed Engl. 42(33), 3871-94. DOI:10.1002/anie.200300605
- Fenn JB et al. (1989) “Electrospray ionization for mass spectrometry of large biomolecules” Science 246(4926), 64-71. DOI: 10.1126/science.2675315
- Tanaka K et al. (1988) “Protein and polymer analyses up to m/z 100 000 by laser ionization time-of-flight mass spectrometry” Rapid Communications in Mass Spectrometry 2(8), 151–153. DOI:10.1002/rcm.1290020802
- Karas M and Hillenkamp F (1988) “Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons” Anal Chem. 60(20), 2299-301. DOI: 1021/ac00171a028
- O’Farrell (1975) “High resolution two-dimensional electrophoresis of proteins” J Biol Chem 250(10), 4007-21.
- Kim MS et al. (2014) “A draft map of the human proteome” Nature 509(7502), 575-81. DOI: 10.1038/nature13302
- Wilhelm M et al. (2014) “Mass-spectrometry-based draft of the human proteome” Nature 509(7502), 582-7. DOI: 10.1038/nature13319
- HUPO: https://hupo.org/human-proteome-project
Egileei buruz: Miren Josu Omaetxebarria, Nerea Osinalde, Jesusmari Arizmendi eta Jabi Beaskoetxea Biokimika eta Biologia Molekularra saileko kideak dira eta Kerman Aloria SGIker-eko teknikaria.
Masa-espektrometriari buruzko artikulu-sorta
- Masa-espektrometria (I). Neoi isotopoetatik elefante hegalariengana
- A new hero is born: Masa-espektrometria justiziaren zerbitzura
- Nor dago icebergaren alden ezkutuan?
- Konposatu galduaren bila
- Metabolomika: osotasuna, zatien baturaren aurrean
- Esploratu gabe dauden lurraldeak kartografiatzen: masa-espektrometria bidezko irudia
- Proteomika: Proteinak eta Masa Espektrometria eskutik helduta
